产品分类
- ZYMO
- ZYMO
- brand
- 普兰德
- 美国Beckman Coulter公司
- Allegra X-15R台式冷冻离心机
- Allegra 64R高速冷冻台式离心机
- Allegra X-30/30R台式高速离心机
- Allegra 25R台式高速冷冻离心机
- Microfuge12/12R台式离心机
- Microfuge 20/20R
- Microfuge 16高速台式离心机
- Avanti J-E多用途高效离心机
- Avanti J-30I高效离心机
- Avanti J-HC大容量离心机
- 新一代Avanti JXN-26智能型高效离心机
- 新一代Avanti JXN-30智能型高效离心机
- Avanti J-26XP系列高效离心机
- J6-MI大容量冷冻离心机
- Optima™XPN超速离心机
- Optima™ XE超速离心机
- Optima MAX-TL台式超速离心机
- Optima™ MAX-XP超速离心机
- Vi-CELL XR细胞存活率分析仪
- Qiyunbio
- 北京百泰克
- 德国Miccra 公司
- 德国Memmert公司
- 海乐枫公司纯水与超纯水系统
- 唯地感受态细胞
- 测序服务
- 杭州米欧仪器
- 美国BD公司流式细胞仪
- 美国Zealway公司高温高压灭菌锅
- 弗德
- 英国Ruskinn公司厌氧、微需氧工作站
- Bio-Rad伯乐
- Genstar 产品
- Union 永联
- 新加坡ESCO公司生物安全柜、CO2培养箱
- 实验仪器
- 实验耗材
- 瑞士Infors公司
- Celltron磁力驱动
- Thermotron振荡培养箱
- Minitron-Bacteria振荡培养箱
- Multitron-Bacteria恒温振荡培养箱
- Minitron Cell振荡培养箱
- Multitron Cell全功能细胞培养箱
- RC落地摇床
- Multitherm摇床
- Multitron Standard系列摇床
- Ecotron摇床
- Orbitron 摇床
- Labfors 5 Cell细胞生物反应器
- Techfors Cell大型生物反应器
- Techfors-S Cell中试原位灭菌生物反应器
- Multifors Cell平行生物反应器
- Labfors Lux Cell光照发酵罐
- Minifors Cell细胞发酵罐
- Minifors Bacteria实验台式发酵罐
- Labfors 5 Bacteria 实验发酵罐
- Labfors Lux光照发酵罐
- Multifors Bacteria 平行发酵罐
- Techfors-S Bacteria中试型发酵罐
- Terrafors Bacteria固态样品发酵罐
- Techfors大型发酵系统
- X-Controlle发酵体系控制系统
- CELLine膜培养瓶
- 实验技术服务
- 武汉优尔生 cloud-clone ELISA kit
- CST
- Omega
- Abcam
- 常用试剂
- Abmart 抗体
- 阿拉丁
- Fermentas
- Invitrogen
新闻中心
联系我们
- 地 址:深圳市南山区西丽街道九祥岭工业区
- 电 话:13006636686,13751058447,0755-33160345
- 传 真:0755-33160345
- 邮 箱:3344179313@qq.com
一、 转录组测序概述
转录组是特定物种、组织或细胞类型转录的所有RNA(转录本)的集合,包括mRNA和非编码RNA(Non-coding RNA, 非编码RNA又包括:tRNA,rRNA,snoRNA,microRNA,piRNA,lncRNA等。通过比较转录组或基因表达谱的研究以揭示生物学现象或疾病发生的分子机制是高通量组学研究的一个常用策略。利用高通量测序技术研究转录组在全面快速得到基因表达谱变化的同时,还可以通过测定的序列信息精确地分析转录本的cSNP(编码序列单核苷酸多态性)、可变剪接等序列及结构变异,另外对于检测低丰度转录本和发现新转录本具有其独特的优势。
二、 转录组测序技术优势
1. 直接得到核酸序列信息,除了得到基因表达量的差异,更可以检测RNA的结构和结构变异。
2. 开放性的转录组分析:无需参考基因组信息,无需设计探针,不但能检测已知基因还能够发现新的转录本。
3. 在测序覆盖率足够大时能够检测到细胞中的低丰度转录本。
4. 随着测序深度的增加可以获得更广的动态检测范围,能够同时鉴定和定量高丰度转录本和低丰度转录本。
三、研究内容
基因表达水平研究:基因表达是将基因中蕴含的遗传信息通过转录、剪接、翻译等转变成功能产物的所有加工过程,是生物生命活动的基础和关键。基因从DNA 转录成RNA 是基因表达的首要步骤。基因转录形成的RNA 丰度不同很大程度地影响到基因最终功能产物的分子浓度。因此,检测基因在RNA水平的表达量即基因转录得到的RNA 的丰度成为人们研究基因表达的关键方法。

研究流程示例
转录本的结构及结构变异研究:利用单碱基分辨率的RNA-Seq 技术可极大地丰富基因注释的很多方面, 包括5′/3′边界鉴定、UTRs区域鉴定以及新的转录区域鉴定等。在发现序列差异(如融合基因鉴定、编码序列多态性研究)方面, RNA-Seq 也展示了其很大的潜力。

研究流程示例
四、 转录组测序实验技术路线

样品要求(RNA)
样品纯度:OD
260/280值应在1.9~2.2 之间,RNA
28S:18S≥1.5,推荐
RIN≥7;DNA 应该去除干净。
样品浓度:最低浓度不低于100ng/µl。
样品总量:每个样品总量不少于7µg。
样品溶剂:要溶解在H20或TE (pH 8.0)中。
样品运输: RNA用冻存管保存,并用干冰或液氮运输。
数据分析技术路线及数据分析内容(无参考基因组)

1. 数据预处理
目的:对原始测序数据进行一定程度的过滤。
原理:根据测序接头以及测序质量对原始的测序数据进行预处理,其中,测序质量Q与测序错误E之间的关系如下:

结果:对预处理后质量以及碱基分布统计进行统计

2. UniGene拼接
目的:将预处理后reads进行拼接,得到拼接结果。
原理: 应用 de
Bruijn graph path 算法对reads进行de novo拼接;对上一步的拼接结果,再用Hamilton
Path算法拼接。
结果:UniGene序列,UniGene统计信息,序列长度分布图

3. 数据库注释
目的:对拼接得到的UniGene进行功能注释
原理:通过blast+算法将拼接得到的UniGene序列与数据库进行比对
结果:比对结果表格,物种分布统计和Evalue分布统计

4. UniGene表达分析
目的:UniGene定量分析。
原理:以UniGene为reference,分别将每个样本的reads进行reference mapping ,从而得到每个样本在每个UniGenes中的一个reads覆盖度,然后应用RPKM/FPKM标准化公式对富集片段的数量进行归一化。


UniGene表达分布图,1X,5X分别为FPKM=1,FPKM=5分界点,可以大体观察到低表达,中表达以及高表达的比例关系

UniGene样本间表达相关性散点图 样本间表达差异程度的MA图,可以体现差异表达总体偏差
5. UniGene表达差异分析
目的:对定量结果进行统计检验分析,找出差异表达UniGene
原理:双层过滤筛选差异基因
FC值筛选:采用Fold-change(FC),表达差异倍数进行第一层此的差异基因筛选
FDR检验:一般采用卡方检验中的fisher精确检验进行p值检验,采用Benjamini FDR(False discovery ratio)校验方法对p值进行假阳性检验,即,通过FDR显著性参数进行第二层次的差异基因筛选。
结果展示:


6. GO功能分类
目的:利用数据库注释信息将
UniGene进行 GO 功能分类。
原理:利用数据库的注释结果,应用blast2GO算法进行GO功能分类,得到所有序列在Gene
Ontology 的三大类:molecular
function, cellular component, biological process 的各个层次所占数目,一般取到14层。
结果:MF,BP,CC三大分类结果文件以及 UniGene2GO 关系列表,三大类别中第二层次上的柱状分布图和饼图,GO功能的层次分布图。



7. KEGG代谢通路分析
目的:对拼接得到
UniGene 进行
KEGG pathway 映射。
原理:应用KEGG
KAAS在线
pathway比对分析工具对拼接得到的UniGene进行KEGG映射分析。
结果:标记的Pathway通路图。

8. COG注释
目的:对拼接得到 UniGene 进行 COG功能分类。
原理:利用blast+算法将拼接得到的UniGene与CDD库中的COG/KOG库进行比对,进行COG功能分类预测,将其映射到COG分类中。
结果: COG分类分布情况图。

9. SSR重复序列注释
目的:对拼接得到 UniGene进行 SSR 简单重复序列的查找。
原理:筛选标准:单核苷酸重复的次数在10次或10次以上,二核苷酸重复的次数在 6次或6次以上,三至六核苷酸重复的次数在 5次或 5次以上。同时,也筛选中间被少数碱基 (间隔小于100或等于100)打断的不完全重复的SSR。
结果:重复序列的信息文件以及统计文件。
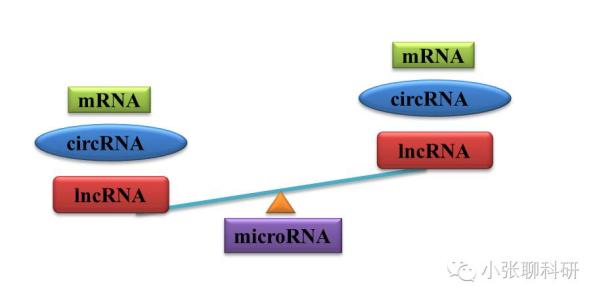
10. LncRNA预测
目的:对拼接得到的UniGene进行LncRNA(Long noncoding RNA)预测。
原理: 通过以下过程对UniGene进行过滤,最终得到候选LncRNA序列。
1) Unigene length > 200bp;
2) Unigene ORF(Open Reading Frame) length < 300;
3) 将满足长度条件的UniGene与多个近源物种进行进化分析,得到序列的保守性和进化特性;
4) 根据上述的特性和已知数据库中coding、noncoding区域的特性建立编码筛选模型;
5) 将符合noncoding模型的UniGene与Pfam等蛋白域数据库进行同源性比对,进一步去除可能的编码特性,最终得出LncRNA预测结果。
数据分析技术路线及数据分析内容(有参考基因组)

1. 数据预处理
目的:对原始测序数据进行一定程度的过滤。
原理:根据测序接头以及测序质量对原始的测序数据进行预处理,其中,测序质量Q与测序错
误E之间的关系如下:


2. 比对基因组
目的:将经过预处理的测序数据与参考基因组进行相似性比对。
原理:Burrower-Wheeler转换算法与splicing比对算法。
1)Burrower-Wheeler转换算法:由于测序数据量非常大,与整条基因组比对所需资源与时间是较为巨大的。目前,我们采用Burrower-Wheeler(BWT)算法对基因进行建立索引、碱基压缩等过程,这样可以很大程度上加快比对速度,减少比对过程中所需资源。
2)splicing比对算法:即分段比对算法,当某条测序序列位于转录本剪切位点时,也就是这条序列同时属于两个外显子,如果将它与参考基因组进行比对,由于基因组两个外显子之间含有intron区,那么它将无法找到它合适的位置;但是应用分段比对算法就可以将这条测序序列分割变成多段子序列,然后应用这些段子序列与基因组进行比对,这样就可以找到它们真正的位置。



3. 基因表达水平研究
目的:应用基因组比对结果进行基因定量。
原理:从指定物种基因模型(基因结构)中得到gene、exon、intron以及UTR等位置信息,通过
基因组比对结果计算出在不用区域富集片段数目,然后应用RPKM/FPKM标准化公式对富集片
段的数量进行归一化。


4. 差异表达分析
目的:应用统计学方法对基因在样本间的表达差异进行分析。
原理:双层过滤筛选差异基因。
FC值筛选:采用Fold-change(FC),表达差异倍数进行第一层此的差异基因筛选。
FDR检验:一般采用卡方检验中的fisher精确检验进行p值检验,采用Benjamini FDR(False discovery ratio)校验方法对p值进行假阳性检验,即,通过FDR显著性参数进行第二层次的差异基因筛选。
结果展示:

5. 转录本结构分析
目的:侦测不同类型的可变剪切事件。
原理:通过测序序列的splicing事件来侦测可能发生剪切连接的候选exon,通过已有可变剪切方式进行验证,最终得出真实的可变剪切事件。
结果展示:对常见的可变剪切方式进行统计分析。

6. 新转录本预测
目的:预测antisense transcript以及intron
transcript。
原理:通过测序序列在基因组上富集的方向性进行反义转录本预测,如果有富集区域方向与基
因转录本方向相反且达到一定的富集阈值,即可认为其为antisense transcript。将完全位于
intron区的一段富集片段作为intron transcript。
7. 新基因预测
目的:预测intergenic区可能存在的新基因并对新基因进行功能注释。
原理:首先,得到在基因间区有测序序列富集的一些段区域;然后,排除那些已经有注释的那些段区域作为候选的新基因。
结果展示:

8. 基因融合分析
目的:寻找可能发生融合功能的基因
原理:通过测序片段的splicing事件以及pair-end测序的距离信息进行基因融合位点的定位,
如果一个测序片段的一个子片段与geneA匹配,另一子片段与geneB匹配,那么geneA与geneB有可能为一个融合基因,而当pair-end双向测序时,一对测序片段中一个与geneA匹配,另一个与geneB匹配,那么geneA与geneB有可能为一个融合基因。如果同时满足两个条件,那么融合发生的可能性就较大。结果展示见右图。

9. GO富集分析
目的:对差异基因相关GO功能进行富集分析。

显著富集GO功能图形统计
10. KEGG富集分析
目的:对差异基因进行KEGG通路富集分析。
原理:应用物种自己的KEGG
pathway进行富集分析,富集结果更加贴近物种现实功能实现的通路,尤其对目前功能注释尚不完全的物种,如,大豆、玉米、葡萄、杨树、白菜、牛、羊等物种的KEGG通路分析。

11. cSNV查找
目的:在转录水平找出变异位点或者片段。
原理:通过测序数据得到基因组每个位点的碱基富集情况;然后,统计每个点富集富集的碱基
种类,得出可能存在的变异(即,与参考基因组碱基不同且富集程度较高的碱基类别)。
结果展示:

12. LncRNA预测
目的:对新转录本进行LncRNA(Long
noncoding RNA) 预测。
原理:通过以下过程对新转录本进行过滤,最终得到候选LncRNA序列:
1) 通过基因组比对得到4类新转录本:Intergenic transcript、Full intron transcript、Antisense transcript、Overlapped with known transcript,将这些新转录本用于LncRNA预测;
2) New Transcript length > 200bp;
3) New Transcript ORF(Open Reading Frame) length < 300;
4) 将满足长度条件的New
Transcript与多个近源物种进行进化分析,得到序列的保守性和进化关系;
5) 根据上述的特性以及已知数据库中coding、noncoding区域的特性建立编码筛选模型;
6) 将符合noncoding模型的New Transcript与Pfam等蛋白域数据库进行同源性比对,进一步去除可能的编码特性,最终得出LncRNA预测结果。
13. LncRNA保守度分析
1)
lncRNA 序列保守性分析lncRNA
序列保守性分析
基于multiple
genome alignment进行序列保守性分析。lncRNA的序列保守性用其序列比对的identity来表示。

2) lncRNA结构保守性分析
通过使用RNAz软件,推断基于多序列比对的一致结构,比较二级结构自由能与一致结构的自由能,得到每一段aligned
region 的SCI 值,并以此作为二级结构的表征,SCI 越大则结构越保守。

3) LncRNA二级结构预测
根据lncRNA的序列保守性,使用RNAalifold软件,可以同时看到其序列比对结果和保守的二级结构。